Neue Methode zur automatisierten Diagnose diabetesbedingter Augenkrankheit
Deep Learning in Kliniken
Künstliche Intelligenz und Deep Learning sind in den letzten Jahren für Kliniken immer interessanter geworden – beispielsweise zur automatisierten Auswertung medizinischer Bilddaten. Um Deep-Learning-Algorithmen beizubringen, Bilder korrekt auszuwerten und Diagnosen vorherzusagen, sind große Mengen an annotierten (mit Informationen versehenen) Daten notwendig.
In Kliniken sind annotierte Daten jedoch oft rar, da das Hinzufügen der Informationen durch Fachkräfte sehr kostspielig ist. In der Forschung war man deshalb auf der Suche nach Lösungen, um den Bedarf an großen Datenmengen mit teuren Annotationen zu reduzieren, ohne Einbuße in der Leistung des Algorithmus in Kauf nehmen zu müssen.
Anwendungsfall diabetische Retinopathie
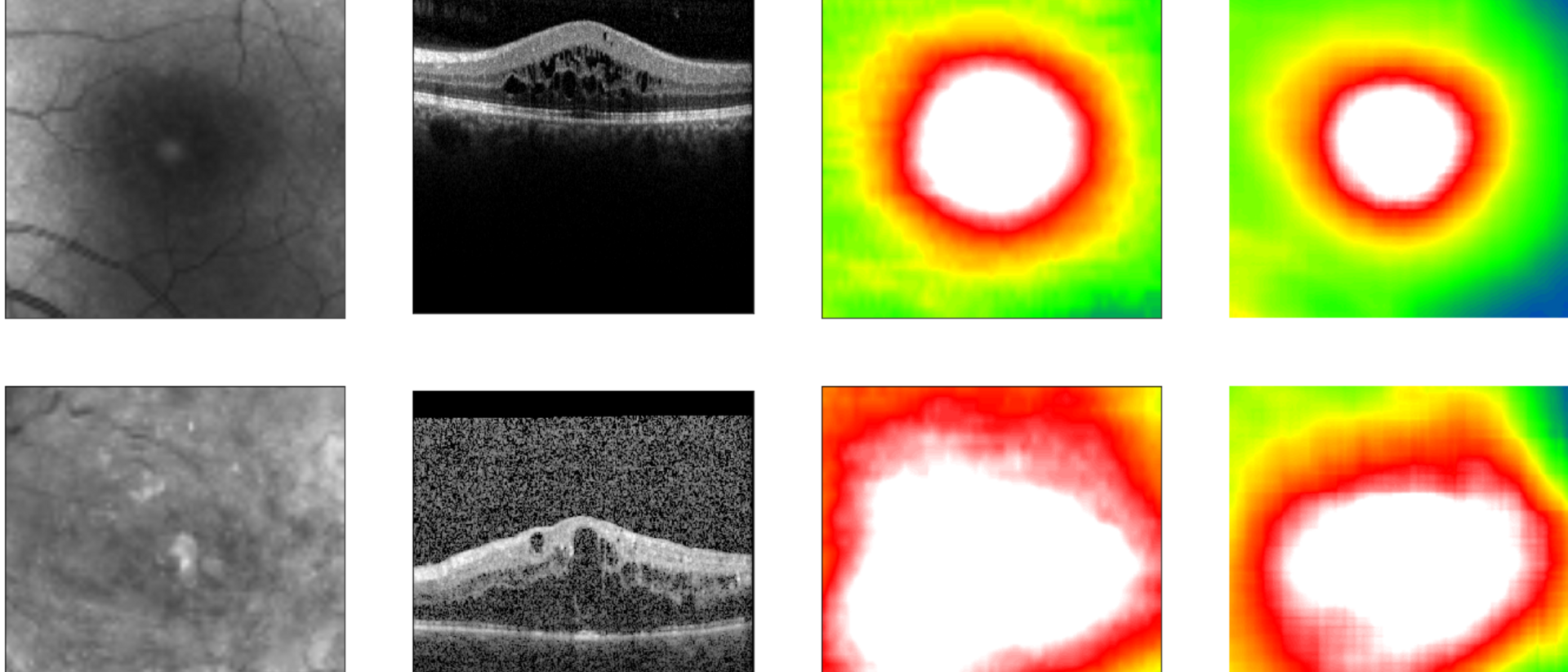
Diabetische Retinopathie ist eine durch Diabetes hervorgerufene Augenerkrankung, die die Netzhaut schädigt und zur Erblindung führen kann. Zur frühzeitigen Diagnose der Krankheit in Risikopatienten wird die Dicke der Netzhaut regelmäßig gemessen. Dafür nehmen Kliniken ein Bild des Fundus auf - von der Oberfläche des Augenhintergrunds. Um die Auswertung dieser Aufnahmen zu automatisieren, haben einige Kliniken begonnen, Deep-Learning-Algorithmen einzusetzen. Diese Algorithmen benötigen eine große Anzahl an Fundus-Bildern mit teuren Annotationen. Anhand dieser lernen sie, die Bilder korrekt auszuwerten.
Die Augenklinik der Ludwig-Maximilians-Universität München (LMU) verfügt über einen Datensatz mit mehr als 120.000 Fundus- und dazugehörigen OCT-Bildern. OCT (optische Kohärenztomografie) liefert präzise Informationen über die Netzhautdicke, wird aber aufgrund der hohen Kosten nicht in jedem Augenzentrum durchgeführt. Die LMU stellte ihre Daten Forschenden der TUM und des Helmholtz Zentrums München zur Verfügung, die auf dem Gebiet der künstlichen Intelligenz in medizinischen Anwendungen Pionierarbeit leisten.
Pre-Training unter „Selbstaufsicht“
„Unser Ziel war es, diese einzigartig große Menge an Fundus- und OCT-Bildern zu nutzen, um eine Methode zu entwickeln, die den Bedarf an teuren annotierten Daten für das Training des Algorithmus reduziert“, sagt Olle Holmberg, Erstautor der Studie von der TUM School of Life Sciences.
Die Forschungsgruppe entwickelte eine neue Methode, die sie als „cross modal self-supervised retinal thickness prediction“ bezeichnet. Diese Methode nutzen sie zum „Pre-Training“ des Deep-Learning-Algorithmus mit den Daten der LMU. Dabei brachte sich der Algorithmus selbst bei, nicht-annotierte Fundus-Bilder auf Basis unterschiedlicher OCT-abgeleiteter Profile der Netzhautdicke zu erkennen. Damit gelang es dem Algorithmus, die Netzhautdicke allein auf Basis des Fundus-Bildes vorherzusagen.
Hohe Leistung mit nur einem Viertel der Trainingsdaten
Die neue Methode verringert den Bedarf an teuren annotierten Daten für das Training des Deep-Learning-Algorithmus erheblich. Bei automatisierten Screenings für diabetische Retinopathie erreichte der Algorithmus die diagnostische Leistung sowohl von medizinischen Fachkräften als auch von Algorithmen, die weit mehr Trainingsdaten benötigen.
„Wir haben es geschafft, den Bedarf an annotierten Daten um 75 Prozent zu reduzieren”, sagt Fabian Theis, Professor für Mathematische Modellierung biologischer Systeme an der TUM und Direktor des Instituts für Computational Biology am Helmholtz Zentrum München. „Die geringe Verfügbarkeit annotierter Daten ist eine große Herausforderung für die Medizin. Wir haben es deshalb zu unserem Ziel gemacht, Methoden zu entwickeln, die weniger Daten benötigen und sich dadurch für den klinischen Einsatz eignen. Kliniken können unseren Screening-Algorithmus für diabetische Retinopathie ab sofort nutzen. Dies ist ein perfektes Beispiel dafür, wie wir mit künstlicher Intelligenz den Alltag in der Medizin und somit auch die Gesundheit aller verbessern können.“
Darüber hinaus erreichten die Forschenden eine Reduktion bei der „Größe des Algorithmus“, d.h. der Anzahl seiner Parameter. Die neue Methode ermöglicht bis zu 200 Mal kleinere Algorithmen. Dies wiederum könnte den Einsatz auf mobilen oder eingebetteten Geräten im klinischen Umfeld erleichtern.
Weitere Anwendungen
Neben der diabetischen Retinopathie kann die neue Methode auch für andere Anwendungen genutzt werden – insbesondere für solche mit vielen Daten ohne Annotation durch Fachkräfte. Im Bereich von Augenkrankheiten könnten Screenings für die altersbedingte Makuladegeneration (AMD) von der neuen Methode profitieren.
Holmberg et al., 2020: Self-supervised retinal thickness prediction enables deep learning from unlabeled data to boost classification of diabetic retinopathy. Nature Machine Intelligence, DOI: 10.1038/s42256-020-00247-1
Der Algorithmus ist auf GitHub verfügbar: https://github.com/theislab/DeepRT
Technische Universität München
Corporate Communications Center
- V. Schulz / K. Baumeister
- presse@tum.de
- Teamwebsite
Kontakte zum Artikel:
Prof. Dr. Fabian Theis
Technische Universität München
Professor für Mathematische Modellierung biologischer Systeme
Boltzmannstr. 3, 85748 Garching
Tel.: +49 (89) 289 - 18386
E-Mail: theis@mytum.de