Korrektursoftware erleichtert Quantifizierung der Entwicklungsschritte von Stammzellen
Klare Sicht auf Stammzellentwicklung
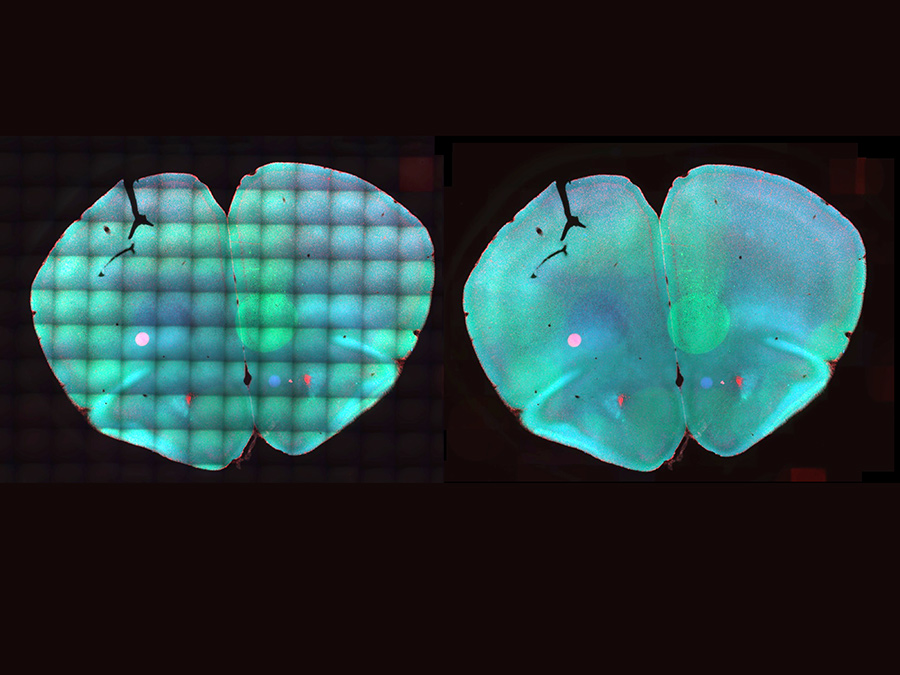
Entwickelt sich eine Stammzelle zu einer spezialisierten Zelle, so geschieht dies in mehreren Schritten. Doch welche Signal-Proteine sind an den entscheidenden Abzweigungen ihres Entwicklungswegs aktiv? Mit der sogenannten Time-lapse microscopy können Forscherinnen und Forscher unter dem Mikroskop einzelne Zellen in sehr hoher zeitlicher Auflösung beobachten und mit Hilfe von Fluoreszenzmarkierungen erkennen, wann genau welches dieser Proteine in der Zelle auftaucht.
Eine einmal identifizierte Stammzelle wird dann mittels Zelltracking-Software über mehrere Tage hinweg quasi auf Schritt und Tritt beobachtet. Diese „Beschattungsarbeit“ gestaltet sich für die Stammzellforscher jedoch oft schwierig. „Die Bilddaten sind oft von unregelmäßiger Helligkeit und einem Verblassen des Hintergrunds im Zeitverlauf gestört“, erklärt Dr. Tingying Peng, Mitarbeiterin der Arbeitsgruppe Quantitative Single Cell Dynamics am Institute of Computational Biology (ICB) des Helmholtz Zentrum München. „Dadurch werden Faktoren, die bei der Entscheidung der Zelle für eine Entwicklungsrichtung auschlaggebend sind, oft nicht oder nur schlecht erkannt.“
Zwar gibt es Algorithmen, die diese Störungen herausfiltern, jedoch benötigen diese entweder extra angefertigte Referenzbilder, eine große Anzahl an Bildern pro Datensatz oder erfordern komplexe manuelle Einstellungen. Zudem korrigiert keine der bestehenden Methoden die Veränderung des Hintergrundes über die Zeit, was die Quantifizierung einzelner Zellen erschwert.
Ein Algorithmus eliminiert Hintergrundveränderungen
Nun stellt Dr. Tingying Peng zusammen mit Dr. Carsten Marr und Professor Nassir Navab, Inhaber des Lehrstuhls für Informatikanwendungen in der Medizin und Augmented Reality an der TU München, einen Algorithmus vor, der die Störfaktoren mit wenigen Bildern pro Datensatz korrigiert.
Die Software trägt den Namen „BaSiC“ und ist frei verfügbar. Sie ist für viele im Bereich Bioimaging verwendete Bildtypen anwendbar, darunter auch für Mosaikbilder, die aus vielen kleinen Einzelbildern bestehen und beispielsweise zur Darstellung von großen Gewebeschnitten verwendet werden. „Vor allem aber“, erklärt Dr. Peng, „kann BaSiC Hintergrundveränderungen in Zeitraffervideos korrigieren. Dies macht es zu einem interessanten Werkzeug für Stammzellforscher, die das Auftauchen bestimmter Faktoren frühzeitig erkennen wollen.“
Das Wesentliche sichtbar machen
Wie gut das neue Korrekturprogramm die Analyse der einzelnen Entwicklungsschritte von Stammzellen verbessert, zeigten die Wissenschaftler am Beispiel von Zeitraffervideos von Blutstammzellen. Mit diesen hatten sie Zellen über sechs Tage hinweg beobachtet. Zu einem bestimmten Zeitpunkt dieser Beobachtungsspanne entscheidet sich eine undifferenzierte Vorläuferzelle für eine von zwei möglichen Entwicklungslinien, die sich danach zu unterschiedlichen reifen Blutzellen fortentwickeln.
Auf den mit BaSiC korrigierten Bildern konnten die Forscher zum Zeitpunkt der Aufspaltung die deutliche Zunahme der Intensität eines bestimmten Transkriptionsfaktors in einer der beiden Zelllinien feststellen, während die Menge des Markers in der anderen Zelllinie gleich blieb. Ohne die vorherige Aufbereitung der Bilder jedoch zeigte sich dieser Unterschied nicht.
„Mit BaSiC konnten wir hier wichtige Entscheidungsfaktoren sichtbar machen, die sonst vor lauter Störungen unsichtbar geblieben wären“, sagt Nassir Navab. „Das Fernziel dieser Forschung ist es, die Entwicklung von Stammzellen gezielt beeinflussen zu können, um beispielsweise für Infarktpatienten neue Herzmuskelzellen züchten zu können. Die neuen Beobachtungsmöglichkeiten bringen uns diesem Ziel ein Stück weit näher.“
Das Korrekturprogramm BaSiC entstand aus einer engen Zusammenarbeit der Lehrstühle für Mathematische Modellierung biologischer Systeme und Informatikanwendungen in der Medizin und Augmented Reality der Technischen Universität München mit dem Institute of Computational Biology (ICB) des Helmholtz Zentrums München. Weiterhin beteiligt waren das Department of Biochemistry and Biophysics der University of California in San Francisco (USA), sowie das Department of Biosystems Science and Engineering (D-BSSSE) der ETH Zürich und der Lehrstuhl für Computer Aided Medical Procedure der Johns Hopkins Universität Baltimore (USA).
Publikation
Tingying Peng, Kurt Thorn, Timm Schroeder, Lichao Wang, Fabian J. Theis, Carsten Marr and Nassir Navab. BaSiC: A Tool for Background and Shading Correction of Optical Microscopy Images.
Nature Communications 8, 14836 (2017) – DOI: 10.1038/ncomms14836
Kontakt:
Dr. Carsten Marr
ICB Institute of Computational Biology
Helmholtz Zentrum München
Ingolstädter Landstr. 1, 85764 Neuherberg, Germany
Tel.: +49 89 3187 2158 – E-Mail - Web
Prof. Dr. Nassir Navab
Lehrstuhl für Informatikanwendungen in der Medizin und Augmented Reality
Technische Universität München
Boltzmannstr. 3, 85748 Garching, Germany
Tel.: +49 89 289 17057 – E-Mail – Web
Technische Universität München
Corporate Communications Center
- Dr. Andreas Battenberg
- battenberg@zv.tum.de
- presse@tum.de
- Teamwebsite



